Cytoscape是一个开源软件平台,用于可视化分子相互作用网络和生物途径,并将这些网络与注释、基因表达谱和其他状态数据集成。Cytoscape核心发行版提供了一组用于数据集成、分析和可视化的基本功能。 应用程序可用于网络和分子分析、新布局、附加文件格式支持、脚本编写以及与数据库的连接。
目录
Cytoscape 3 新功能
支持多种标准
Cytoscape 支持许多标准网络和注释文件格式,包括:SIF(简单交互格式)、GML、 XGMML、BioPAX、 PSI-MI、 GraphML、 KGML(KEGG XML)、 SBML、OBO和Gene Association。还支持分隔文本文件和 MS Excel™ 工作簿,您可以导入数据文件,例如表达式配置文件或GO由其他应用程序或电子表格程序生成的注释。使用此功能,您可以加载和保存节点、边和网络上的任意属性。例如,为您的蛋白质输入一组自定义注释术语,为您的蛋白质-蛋白质相互作用创建一组置信值。
公共数据库客户端
Cytoscape 作为 Web 服务客户端运行。这意味着Cytoscape可以直接连接到外部公共数据库并导入网络和注释数据。目前,支持Pathway Commons、 IntAct、BioMart和NCBI Entrez Gene 。我们继续为流行数据库开发新的服务客户端。
互操作性
- 由于 Cytoscape 支持导入/导出标准文件格式,因此您可以轻松地将 Cytoscape 放入您的工作流程中。例如,如果您有 igraph或 Bioconductor生成的网络数据,Cytoscape 可以将该文件作为文本表加载,您可以将其以 PSI-MI 格式导出以供其他生物信息学工具或您自己的应用程序/脚本使用。
- 从版本 3.3 开始,Cytoscape 支持RESTful API进行编程访问。您可以使用您选择的编程语言来访问 Cytoscape 核心功能,例如创建网络、应用布局或图像导出。
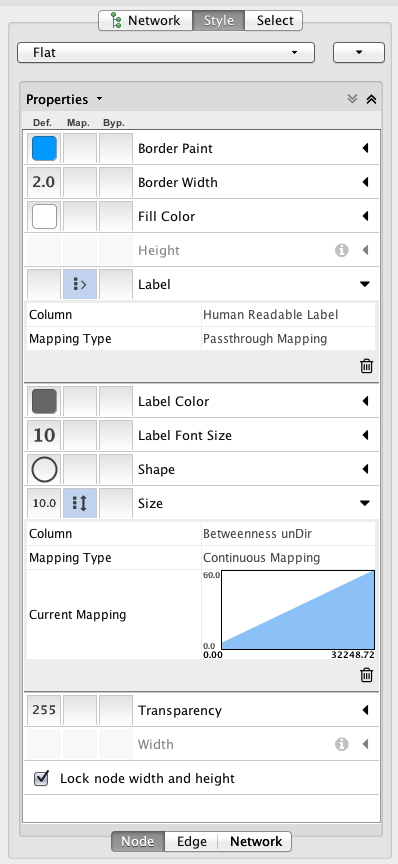
可视化映射器
使用强大的 VisualStyles自定义网络数据显示。查看网络上基因表达比率和 p 值的叠加。 表达数据可以根据用户可配置的颜色和可视化方案映射到节点颜色、标签、边框粗细或边框颜色等。
筛选
过滤网络以根据当前数据选择节点和/或交互的子集。 例如,用户可以根据加载基因表达数据的 p 值,选择参与阈值数量的交互的节点、共享特定 GO 注释的节点、或者基因表达水平在一种或多种条件下显着变化的节点。您可以根据过滤结果创建新网络。
Cytoscape 3.10.3+插件安装教程
这里以windows安装为例
安装 Cytoscape
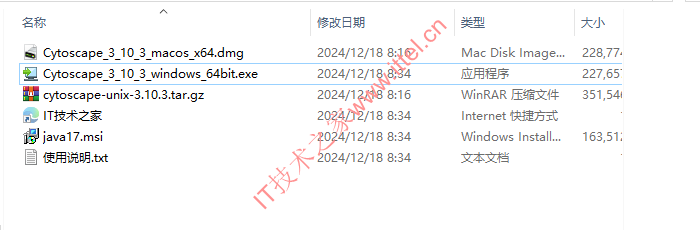
1、下载解压
- windows安装包:Cytoscape_3_10_3_windows_64bit.exe
- Linux安装包:cytoscape-unix-3.10.3.tar.gz
- mac安装包:Cytoscape_3_10_3_macos_x64.dmg
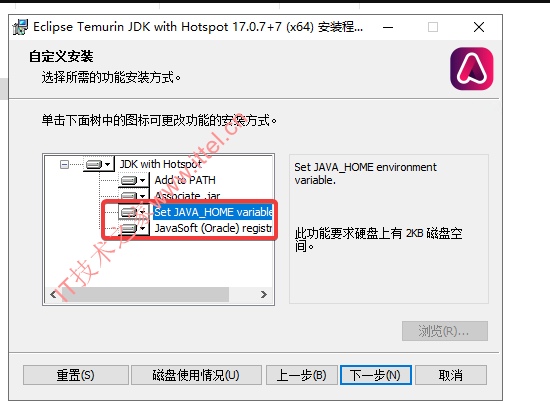
2、安装java17,这两个也要选上
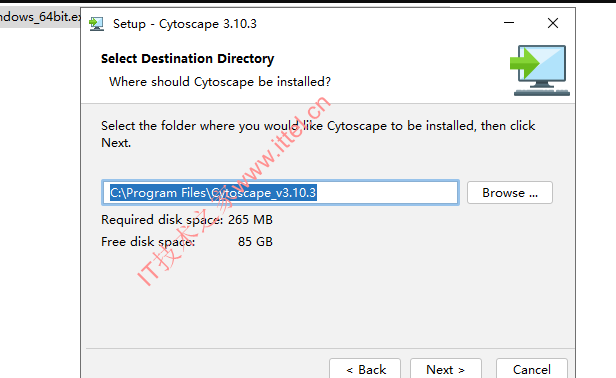
2、安装Cytoscape_3_10_3_windows_64bit
3、安装成功
安装插件
- 确保主程序已经正确安装
- 复制插件到主程序安装路径下的【apps】目录即可,windows默认是:C:\Program Files\Cytoscape_v3.10.3\apps
- Linux目录是:cytoscape目录下的/apps/installed/
- 运行软件,点击APPS 即可使用
相关文件下载
Cytoscape 3.10.3下载
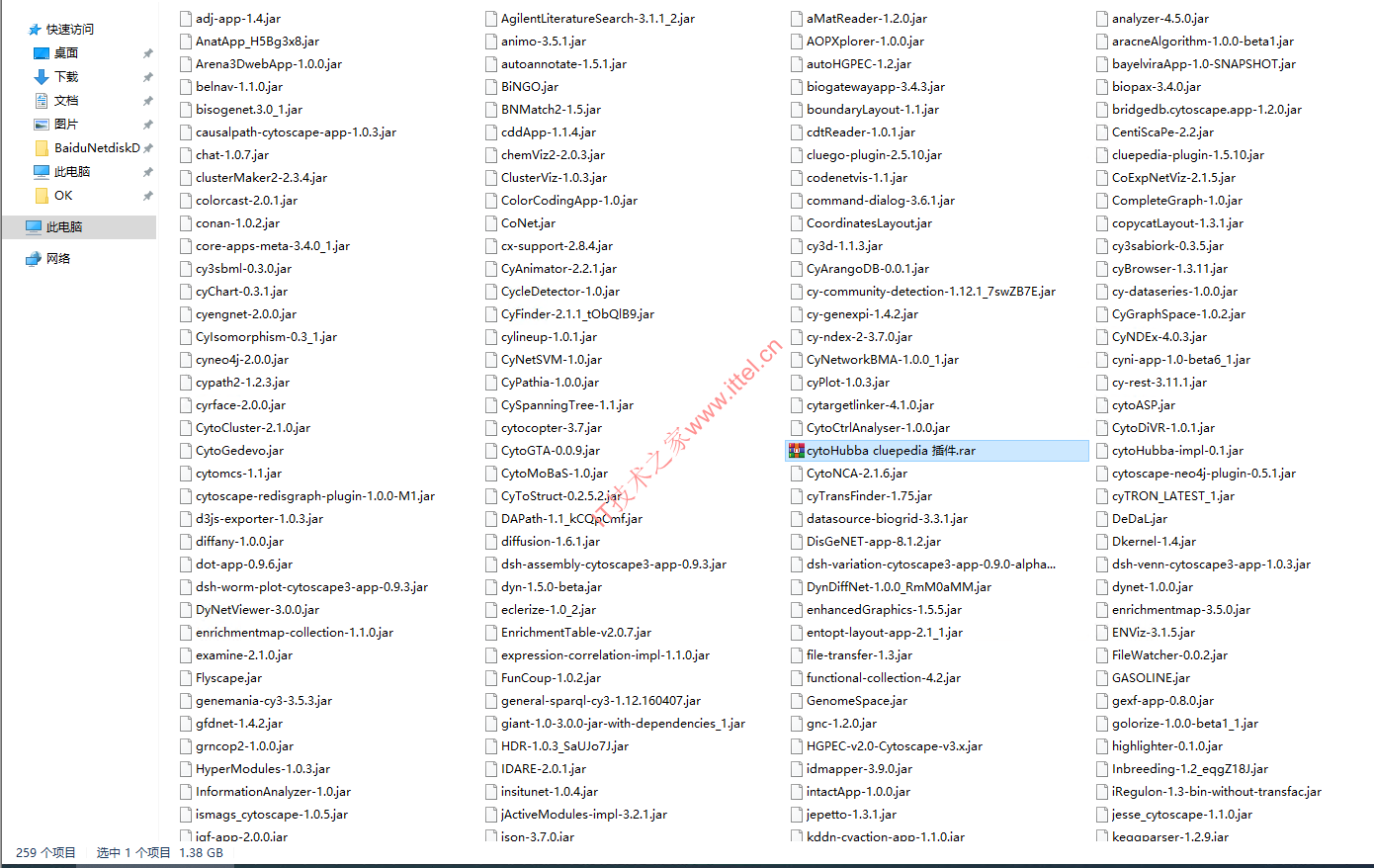
cytoHubba cluepedia离线插件下载
包含258个最新离线插件【2024年12月18日更新】
注:本站统一解压密码为www.ittel.cn


 微信扫一扫打赏
微信扫一扫打赏
 支付宝扫一扫打赏
支付宝扫一扫打赏


安装以后打不开啊,哥
特别java,是不是有其他版本冲突,检查下自己的环境
我没有Java啊,都是按照你上面的安装的
来自Deepseek
1. 检查 Java 环境
Cytoscape 需要 Java 运行时环境(JRE)才能运行。如果未安装或版本不兼容,可能会导致无法启动。
解决方法:
确保已安装 Java 8 或 Java 11(Cytoscape 3.10.3 支持这两个版本)。
访问 Java 官方网站 下载并安装合适的版本。
安装后,检查 Java 是否配置正确:
在终端或命令提示符中运行 java -version,确认版本号。
2. 检查系统权限
如果 Cytoscape 安装目录的权限不足,可能会导致无法启动。
解决方法:
确保你有权限访问 Cytoscape 的安装目录。
尝试以管理员身份运行 Cytoscape:
在 Windows 上,右键点击 Cytoscape 图标,选择“以管理员身份运行”。
3. 检查日志文件
Cytoscape 启动失败时,通常会生成日志文件,其中可能包含错误信息。
解决方法:
找到 Cytoscape 的日志文件:
Windows:C:\Users\<你的用户名>\CytoscapeConfiguration\
打开 cytoscape.log 文件,查看是否有错误信息。